En el proceso de realizar estudios genéticos, a menudo nos encontramos con muestras de ARN insuficientes, por ejemplo, para estudiar pequeños tumores orales anatómicos, incluso muestras de una sola célula y muestras de mutaciones genéticas específicas que se transcriben en niveles muy bajos en células humanas.Eso sí, para la prueba de COVID-19, si los hisopados no están en el lugar correcto o no se dan los tiempos suficientes durante la toma de muestras, el tamaño de la muestra será muy bajo, razón por la cual la Comisión de Salud y Planificación Familiar salió hace dos días y pasó la prueba, y si el muestreador de ácido nucleico no tomó seis muestras, puede informarlo.
La sensibilidad del reactivo es importante porque tenemos este o aquel problema, entonces, ¿qué podemos hacer para mejorar la sensibilidad de la RT-PCR?
Antes de discutir posibles soluciones, mencionemos dos grandes complicaciones con la situación que acabamos de mencionar.
En primer lugar, nos preocupa la pérdida de ARN cuando solo tenemos unas pocas poblaciones de células en nuestra muestra.Si se utilizan métodos tradicionales de separación y limpieza, como el método de columna o el método de precipitación de ácidos nucleicos, existe una gran posibilidad de que se pierdan las pocas muestras.Una solución es agregar una molécula transportadora, como tRNA, pero incluso así, no hay garantía de que nuestro experimento de recuperación esté bien.
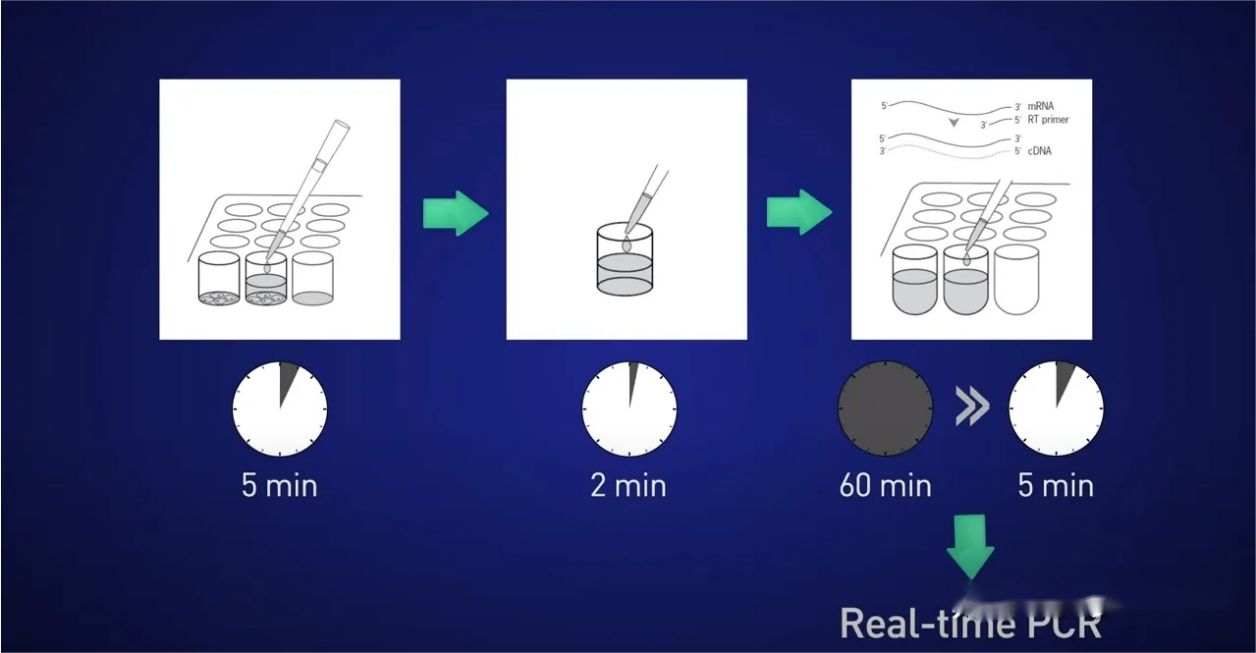
Entonces, ¿cuál es una mejor manera?Una buena opción para células cultivadas o muestras microanatómicas es utilizar lisis directa.
La idea es dividir las células durante 5 minutos, liberar el ARN en la solución, luego detener la reacción durante 2 minutos, luego agregar el lisado directamente a la reacción de transcripción inversa para que no se pierda el ARN y finalmente colocar el ADNc resultante directamente. en la reacción en tiempo real.
Pero, ¿y si, debido a un punto de partida limitado oa una pequeña cantidad de expresión del gen objetivo, podemos reciclar todo el ARN y aun así no proporcionar suficientes plantillas para obtener una buena señal en tiempo real?
En este caso, el paso de preamplificación puede ser muy útil.
El siguiente es un esquema para aumentar la sensibilidad después de la transcripción inversa.Antes de comenzar, debemos preguntarnos qué objetivos nos interesan aguas abajo, a fin de diseñar cebadores específicos para estos objetivos para la preamplificación.
Esto se puede lograr creando una imprimación mixta con hasta 100 pares de imprimaciones y un ciclo de reacción de 10 a 14 veces.Por lo tanto, se necesita una mezcla maestra diseñada específicamente para este requisito para preamplificar el ADNc obtenido.
La razón para establecer el número de ciclos entre 10 y 14 es que este número limitado de ciclos garantiza la aleatoriedad entre los distintos objetivos, lo cual es crucial para los investigadores que necesitan información molecular cuantitativa.
Después de la preamplificación, podemos obtener una gran cantidad de cDNA, por lo que la sensibilidad de detección en el back-end mejora considerablemente, e incluso podemos diluir la muestra y realizar múltiples reacciones de PCR en tiempo real para eliminar posibles errores aleatorios.
Hora de publicación: 11-abr-2023